01.10.2020 Fachübergreifend
„Vernetzen. Forschen. Heilen.“ Deutschlands Weg von der reinen Digitalisierung in die KI

EINBLICK IN EIN PROJEKT ZUR ENTWICKLUNG EINER OFFENEN, FORSCHUNGSKOMPATIBLEN E-HEALTH-PLATTFORM IN DEUTSCHLAND
Warum brauchen wir mehr als nur Digitalisierung?
SARS-CoV-2, Auslöser der COVID-19-Pandemie, ist noch nicht überstanden, doch bereits wenige Monate nach erstmaligem Nachweis des Virus verfügen wir über Detailinformationen zur Herkunft, Epidemiologie, Virusstruktur, Pathogenität und Virulenz zu einem neuen Vertreter einer ansonsten bekannten Virusspezies (Coronaviren). Dies ist möglich, da die Infektionsfolgen als „beispiellose globale Bedrohung“ eingestuft wurden, China erstmals mehr Transparenz in der Surveillance zuließ und eine Vernetzung der Staaten zum Datenaustausch und Abgleich seiner Maßnahmen auf übergeordneten Ebenen (z.B. Europaparlament, CDC, ECDC, WHO u.a.) wahrgenommen wurde.
Allerdings führt die Akuizität des Geschehens, die unablässige mediale Aufmerksamkeit und der politische Regulierungswille zu einem enormen Wissenschafts- und Publikationsdruck, so dass die bisherige Daten- und Studienqualität sehr uneinheitlich ist. Das mittel- und unmittelbare Erleben einer Infektionskrankheit und deren Auswirkungen auf Gesundheit und Gesellschaft hat auch das allgemeine Verständnis für nosokomiale Infektionen (NI) in der Bevölkerung geschärft.
Jedes Jahr erkranken in Deutschland über 500.000 Patientinnen und Patienten während einer Krankenhausbehandlung an einer nosokomialen Infektion, womit sie zu den häufigsten Komplikationen eines stationären Aufenthaltes gehören (1, 2). Die Inzidenz von NIs in Deutschland ist damit mehr als doppelt so hoch wie die aller klassischen Infektionserkrankungen. Dadurch haben sie nicht nur einen erheblichen Einfluss auf den betroffenen Patienten, sondern auch gesellschaftlich und volkswirtschaftlich bedeutsame Effekte (3). Wie Deutschland in der aktuellen Corona Pandemie quasi in Echtzeit erlebt hat, führt das Nichterkennen von Erregerübertragungen oder Clustern zu Ausbrüchen, die oft nur schwer beherrschbar sind (4, 5).
 Berücksichtigt man, dass ein durchschnittliches Krankenhaus jährlich hunderte Terabyte (1 Terabyte = 1012 Byte) an Daten im Rahmen seiner routinemäßigen Patientenversorgung generiert, könnte man annehmen, dass eine Nutzung zu Forschungszwecken raschen Erkenntnisgewinn und entsprechenden medizinischen Fortschritt garantieren müsste.
Berücksichtigt man, dass ein durchschnittliches Krankenhaus jährlich hunderte Terabyte (1 Terabyte = 1012 Byte) an Daten im Rahmen seiner routinemäßigen Patientenversorgung generiert, könnte man annehmen, dass eine Nutzung zu Forschungszwecken raschen Erkenntnisgewinn und entsprechenden medizinischen Fortschritt garantieren müsste.
Bis jetzt versickern die ungeheuren heterogenen Datenmengen in Datensilos von Institutionen und Gesundheitseinrichtungen, denn wesentliche Voraussetzungen für eine wissenschaftliche übergreifende Nutzung fehlen und/oder müssen hierfür noch geschaffen werden:
- Keine einheitlichen Datenmanagement- und Informationssysteme der Krankenhäuser, somit auch keine oder nur vereinzelte standortübergreifende Vernetzung.
- Uneinheitliche Datenqualität (lückenhafte Dokumentation, unterschiedliche Erfassungskriterien und -methoden) erschwert Zusammenführung und Auswertbarkeit (Nutzbarkeit, Interoperabilität).
- Zunehmende Datenschutzauflagen erschweren Klinik- und Institutsübergreifenden Austausch von Patientendaten, auch für Forschungszwecke (translationale Forschung).
- Techniken des sog. maschinellen Lernens (mathematische Rechenprogramme zur biostatistischen Gewichtung von Einzeldaten, Clustering, Algorithmenbildung etc.) zur medizinisch wissenschaftlichen Analyse von großen Datenmengen (BigData) sind nur möglich, wenn solche dafür auch zur Verfügung gestellt werden.
- Keine Vereinheitlichung und Standardisierung von medizinischen Begriffen (Terminologie, Nomenklatur) oder Übersetzung freitextlicher Dokumente (z.B. Freitextbefunde, Arztbriefe) in digital lesbare Codes.
Ein Schatz soll gehoben werden: Nutzbarmachung von Versorgungsdaten
Das Bundesministerium für Bildung und Forschung (BMBF) hat diese Probleme als nationalen Standortnachteil für die medizinische Forschung und Patientenversorgung in Deutschland erkannt und im November 2015 das Förderkonzept „Medizininformatik Initiative (MII): Vernetzen. Forschen. Heilen.“ ins Leben gerufen, für das in einer ersten Stufe 160 Mio Euro Fördergelder zur Verfügung gestellt wurden.
Die Initiative will durch finanzielle Förderung ermöglichen, von einer bislang bloßen Digitalisierung des Gesundheitswesens zu einer darüber hinaus gehenden patientenorientierten wissenschaftlichen Nutzung der Datenmengen im Sinne einer translationalen Forschung zu gelangen.
Am Ende stünde die viel zitierte „Künstliche Intelligenz“, die Ärztinnen und Ärzten standortübergreifend zur Verfügung gestellt werden und behilflich in der Prävention, Diagnostik und Therapie von Erkrankungen sein könnte.
Die Medizininformatikinitiative adressiert alle Universitätsklinika Deutschlands, die sich in vier Konsortien (HiGHmed, MIRACUM, SMITH und DIFUTURE) organisiert haben. Neben den Universitätsklinika sind hier Partner wie Forschungseinrichtungen, Hochschulen, Unternehmen und nicht universitäre Krankenhäuser eingeschlossen, welche die technischen und organisatorischen Voraussetzungen schaffen sollen, Daten aus der Forschung und Patientenversorgung austauschen zu können. Dazu gehört der Aufbau von so genannten Datenintegrationszentren (DIZ), in der die dort eintreffenden Daten sicher verwaltet, lesbar, sortiert und ggf. entsprechend spezifischer wissenschaftlicher Interessen und Anwendungen durch speziell hierfür entwickelte Algorithmen analysiert, ausgewertet und zurückgespiegelt werden können. Die Konsortien verfolgen in Anwendungen, so genannte Use Cases, unterschiedliche wissenschaftliche Fragestellungen.
Beispiele für solche Use Cases sind die IT-basierte Unterstützung von Diagnose und Therapiewahl bei seltenen Erkrankungen, die Rekrutierung von Teilnehmern für klinische Studien, die personalisierte Krebstherapie, die Ausbruchsfrüherkennung oder andere Aufgabenstellungen aus dem Forschungs- und Versorgungsalltag. Mit Hilfe dieser konkreten Anwendungsfälle soll der Mehrwert eines Datenaustauschs und der IT-Lösungen demonstriert werden. Die Konsortien tragen außerdem mit verschiedenen Maßnahmen, z.B. Entwicklung von Ausbildungsprogrammen oder Einrichtung von Professuren und Nachwuchsgruppen dazu bei, die Medizininformatik in Deutschland zu stärken.
Das Konsortium HiGHmed (6) bündelt und integriert die Kompetenzen von acht Universitätsklinika und medizinischen Fakultäten sowie Partnern aus Wissenschaft und Industrie. Es unterteilt sich in drei Anwendungsbereiche oder Use Cases:
Onkologie: Der Use Case Onkologie befasst sich mit der Aufgabe, enorme Datenmengen aus der Genomsequenzierung und Radiologie in die klinische Praxis zu integrieren. Ein virtuelles Onkologiezentrum wird den Behandlungsverlauf von Krebspatienten veranschaulichen und den Kliniken, Forschungseinrichtungen, Ärzten und Patienten als Austauschplattform dienen. So sollen vergleichbare Krebsfälle besser erkannt und eine individuelle patientenorientierte Behandlung ermöglicht werden.
Kardiologie: Neben klinischen Daten werden neue, mobile Diagnosetechnologien für die Langzeitbeobachtung von chronisch Kranken erprobt. Sie ermöglichen es, umfangreiche und mitunter sehr spezifische Daten zu erfassen. Diese Daten können genutzt werden, um bei Patienten mit einem hohen Risikopotential für kardiovaskuläre Erkrankungen frühzeitig Verschlechterungsschübe zu erkennen und somit langfristig Krankenhausaufenthalte und die Sterblichkeitsrate zu verringern.
Infektionskontrolle: Vorrangig soll das Projekt der Verbesserung der Patientensicherheit durch vollständige, personenunabhängige und möglichst automatisierte Integration verschiedener Daten (zum Beispiel medizinische Patienten-, Mikrobiologie- und Patientenbewegungsdaten innerhalb des Krankenhauses) aus diversen Primärsystemen und der Früherkennung von Clustern bakterieller Erreger typischer Krankenhausinfektionen dienen.
Die Daten verschiedener Klinikstandorte sollen über Datenintegrationszentren zusammengefasst und ausgewertet werden. Komplexe Rechenalgorithmen und -systeme ermöglichen die Darstellung der Daten in offenen Informationsmodellen. Angelegt als lernender Algorithmus sollen so aus der Fülle der Daten Cluster erkannt und gewichtet werden. In geeigneter Form visualisiert, können sie zur Häufungs- und Ausbruchsfrüherkennung genutzt werden (Detektieren, Beobachten, Visualisieren, Analysieren, Berichten).
Ziel soll es sein, eine für den medizinischen Anwender lokale benutzerfreundliche Oberfläche zu implementieren, welche ad hoc kumulierte Versorgungsdaten in Echtzeit visualisiert, die Rückverfolgbarkeit ermöglicht und darüber hinaus gestattet, Hypothesen zu Risikokonstellation sowie Interventionen – auch und gerade standortübergreifend – zu generieren.
Die Erkennung identischer Erreger und Vernetzung dieser Informationen mit den Bewegungsdaten der Patienten stellt die Grundlage für eine Muster- und Transmissionsfrüherkennung auch von bakteriellen Erregern ohne besondere Marker-Eigenschaften dar. Ist die vollständige und biomedizininformatische Integration und Analyse komplexer Zusammenhänge erfolgreich, kann eine Früherkennung von Transmissionsereignissen in einem zeitlich räumlichen Kontext erfolgen und somit Ausbrüche verhindert oder erheblich abgeschwächt werden, da Interventionen zeitlich früher initiiert werden können (7, 8) (Abbildung 1).
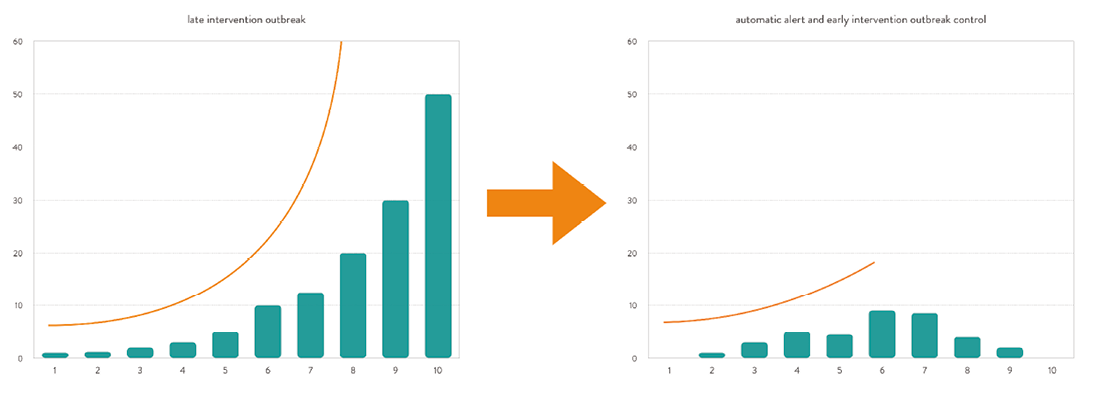
Abb. 1
Den teilnehmenden Konsortialpartnern soll es dann möglich sein, in Echtzeit eine Übersicht über mögliche Transmissionen, Erreger und Erreger-Cluster zu erhalten, um so risikoadjustiert handeln zu können.
Die Medizininformatik Initiative ging 2017 nach Beendigung der Konzept- in eine Aufbau- und Vernetzungsphase über, welche 2021 enden soll.
Innerhalb der Aufbau- und Vernetzungsphase (2018 – 2021) sollen bei den beteiligten Partnern sogenannte Daten-Integrationszentren (DIZ) aufgebaut und übergreifende IT-Lösungen für die jeweiligen Anwendungsbereiche entwickelt werden. In dieser Phase sollen auch Vernetzungsprojekte zwischen den Konsortien und neuen Partnern entstehen. Dazu wurden bereits Gespräche mit potenziellen Standorten und medizinischen Einrichtungen geführt.
Ende 2021 steht eine Auditierung und Evaluierung der einzelnen Konsortien und Anwendungsfälle an.
Falls die Forschung der Phasen die erhofften Ergebnisse erbringen, können die jeweiligen Projekte dann in einer anschließenden Ausbau- und Erweiterungsphase ab 2022 in die breite Anwendung gebracht und auf weitere Standorte deutschlandweit übertragen werden. Hierzu sind weitere Fördermodule des BMBF geplant.
Ein Fazit der von Restriktion und Abstand geprägten COVID-19-Pandemie ist bereits jetzt in aller Munde: die Aufwertung und Wertschätzung digitaler Arbeits- und Kommunikationsmöglichkeiten, deren Chancen, aber auch Limitierungen, wir intensiv erfahren haben und deren Weiterentwicklung dringend notwendig ist.
Weiterführender Link zur Medizininformatik Initiative (MII): https://www.medizininformatik-initiative.de/de/start
Weiterführender Link zu den Konsortien der Initiative: https://www.medizininformatik-initiative.de/de/konsortien
Weiterführender Link zum HiGHmed-Konsortium: https://www.medizininformatik-initiative.de/de/konsortien/highmed
Literatur
[1] Cassini A, Hogberg LD, Plachouras D, Quattrocchi A, Hoxha A, Simonsen GS, et al. Attributable deaths and disability-adjusted life-years caused by infections with antibiotic-resistant bacteria in the EU and the European Economic Area in 2015: a population-level modelling analysis. Lancet Infect Dis. 2019;19(1):56-66.
[2] Geffers C, Sohr D, Gastmeier P. Mortality attributable to hospital-acquired infections among surgical patients. Infect Control Hosp Epidemiol. 2008;29(12):1167-70.
[3] Cassini A, Plachouras D, Eckmanns T, Abu Sin M, Blank HP, Ducomble T, et al. Burden of Six Healthcare-Associated Infections on European Population Health: Estimating Incidence-Based Disability-Adjusted Life Years through a Population Prevalence-Based Modelling Study. PLoS Med. 2016;13(10):e1002150.
[4] Sejdić E. Medicine: Adapt current tools for handling big data. Nature. 2014;507(7492):306.
[5] von Landesberger T. Entwicklung einer Visualisierung im Use Case Infection Control. HiGHmed Vollversammlung, TU Darmstadt, 2018.
[6] Haarbrandt B, Schreiweis B, Rey S, Sax U, Scheithauer S, Rienhoff O, et al. HiGHmed – An Open Platform Approach to Enhance Care and Research across Institutional Boundaries. Methods Inf Med. 2018;57(S 01):e66-e81.
[7] Steele L, Orefuwa E, Dickmann P. Drivers of earlier infectious disease outbreak detection: a systematic literature review. Int J Infect Dis. 2016;53:15-20.
[8] Swaan C, van den Broek A, Kretzschmar M, Richardus JH. Timeliness of notification systems for infectious diseases: A systematic literature review. PLoS One. 2018;13(6):e0198845.
Weitere Literatur auch unter https://www.medizininformatik-initiative.de/de/ueber-die- initiative/publikationen
Seifert J, Scheithauer S, Eils R, Stengel D, Ekkernkamp A: „Vernetzen. Forschen. Heilen.“: Deutschlands Weg von der reinen Digitalisierung in die KI. Passion Chirurgie. 2020 Oktober, 10(10): Artikel 05_02.
Autoren des Artikels
Prof. Dr. med. Julia Seifert
Präsidiumsmitglied im BDCStellv. Leiterin Deutsche Akademie für Fort- und Weiterbildung des BDCABS-Expertin und Hygienebeauftragte ÄrztinVorsitzende Interdisziplinäre Sektion Krankenhaus- und Praxishygiene der DGKHKlinik für Unfallchirurgie und Orthopädie, Unfallkrankenhaus Berlin kontaktieren
Prof. Dr. med. Axel Ekkernkamp
Ärztlicher Direktor und GeschäftsführerUnfallkrankenhaus Berlin (ukb)Warener Str. 712683Berlin kontaktierenWeitere aktuelle Artikel
09.05.2025 BDC|News
Update zur Wundversorgung
Nahezu 1 Million behandlungsbedürftige Fälle von chronischen oder schlecht heilenden Wunden, Kosten für das Gesundheitssystem von geschätzt 8 Mrd. Euro werden als Belastung für das deutsche Gesundheitswesen bezeichnet.
01.05.2025 Fachübergreifend
Die Rolle der Chirurgie in der Versorgung chronischer Wunden
Das deutsche Gesundheitssystem steht in den nächsten Jahren vor immensen Herausforderungen. Die politisch gewünschte Zentralisierung und hieraus folgende Reduzierung stationärer Leistungserbringer sowie die Verschiebung von Behandlungen in den ambulanten Sektor wird auch für die Versorgung von Menschen mit chronischen Wunden zu noch nicht absehbaren Verwerfungen und vermutlich auch zu einer Verschlechterung der Behandlungsqualität, der ohnehin oftmals bereits unterversorgten Patienten führen.
01.05.2025 Fachübergreifend
Was benötigen spezialisierte Leistungserbringer zur Behandlung schwer heilender und chronischer Wunden von Chirurg:innen für eine gute Zusammenarbeit
Wunden haben immer eine Ursache. Sie sind Folge eines Ereignisses oder Symptom einer Erkrankung. Interne Faktoren, wie zum Beispiel Erkrankungen der Atemwege, des Verdauungs-, Herz-Kreislauf-, des Immunsystems oder Funktionsverlust der Haut können zusätzlich die physiologischen Mechanismen der Wundheilung stören.
01.05.2025 Fachübergreifend
Delegation von Wundmanagement an nicht-ärztliches Personal
Sowohl in Kliniken als auch bei vertragsärztlich niedergelassenen Leistungserbringern* stellt sich, insbesondere angesichts der Personalsituation und der stetig steigenden Zahl an Patienten, immer wieder die Frage der zulässigen Delegation von ärztlichen Leistungen an nicht-ärztliches Personal.
Lesen Sie PASSION CHIRURGIE!
Die Monatsausgaben der Mitgliederzeitschrift können Sie als eMagazin online lesen.